Sistema del Complemento (C')
Paralelamente a la barrera de los agentes antimicrobianos, entra en juego el sistema del complemento.
Consta de un gran número de proteínas plasmáticas solubles, localizadas en sangre u otros fluidos, que interactúan tanto para opsonizar patógenos como para inducir la respuesta inflamatoria contra la infección. Muchas de estas proteínas son proteasas que se activan tras su división por otras proteasas específicas.
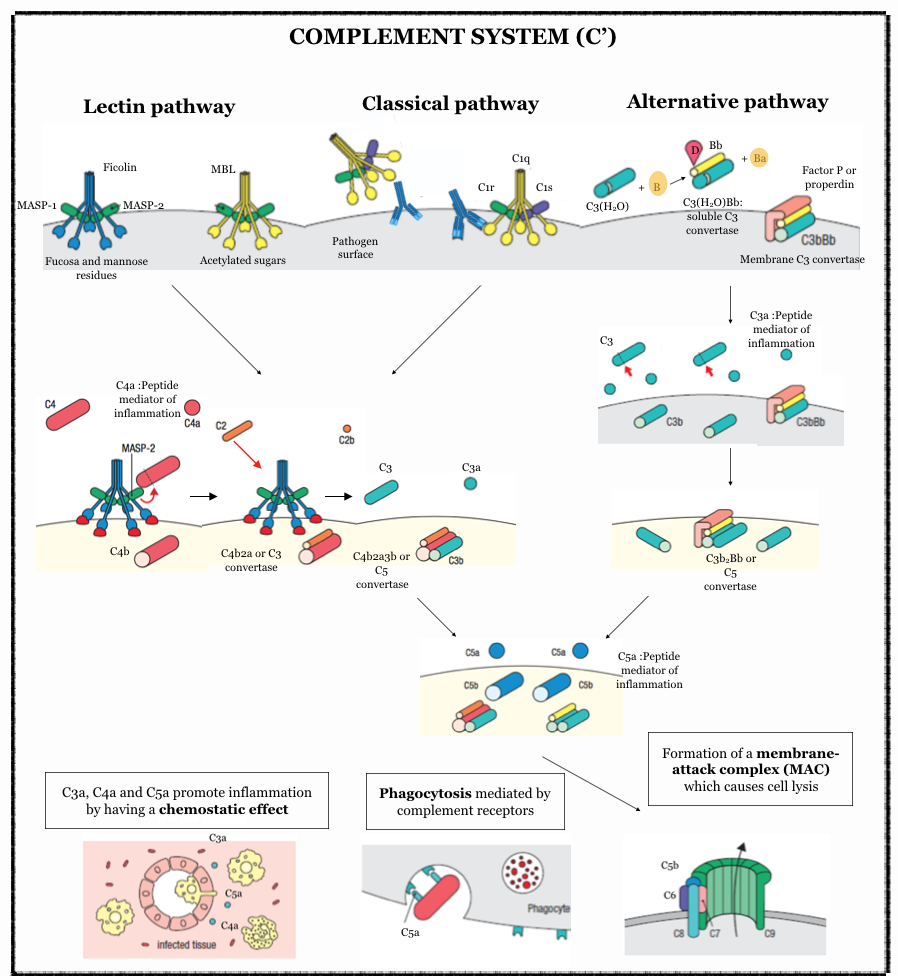
Este sistema puede activarse por tres vías:
- Vía clásica: es la más reciente evolutivamente, y la primera en ser descubierta.
- Vía alternativa:es la más antigua evolutivamente, descrita tras la vía clásica.
- Vía de las lectinas:ocupa un lugar intermedio en la evolución, y es la más reciente en ser caracterizada.
Independientemente de la vía que se siga, la activación del complemento consta de una serie de fases:
- Fase de reconocimiento (3 vías): Es una fase muy delimitada en la vía de las lectinas y en la vía clásica. En la vía alternativa la situación es algo diferente ya que la activación se produce de forma intrínseca sin reconocimiento previo.
- Generación de la C3 y C5 convertasa: mediante una cascada enzimática mediada por zimógenos.
- Formación del complejo de ataque a la membrana: MAC (membrane attack complex) y la C5b forman un poro en la membrana que lisa la bacteria.
NOTA: A partir de ahora, se hará uso de una nomenclatura en la que el fragmento mayor va a asociado a la letra b (C4b, C3b y C5b) salvo en el caso de C2, en el que C2a es el fragmento mayor.
Vía alternativa
La característica principal de está vía es que de forma espontánea el factor C3, que presenta alta afinidad por el agua, se une a una molécula de agua, permitiendo la unión del factor B en fase soluble.
A continuación una proteasa especial de la vía, llamada factor D, se une al complejo C3(H2O)B y escinde al factor B en dos fragmentos liberándose el fragmento Ba y formándose la convertasa C3 soluble llamada C3(H2O)Bb.
Esta C3 convertasa soluble, una proteasa de residuos de serina, va a actuar sobre moléculas de C3, dando lugar a C3a y C3b. La acción proteolítica reside en el elemento Bb. El C3b quedará unido a la superficie bacteriana mediante un enlace tioéster (actuando a modo de marcador para la opsonización) mientras que el C3a liberado tendrá acción quimiotáctica.
C3b en la membrana se une de nuevo al factor B, que tras ser escindido por el factor D y liberar Ba, formará el C3bBb (C3 convertasa de membrana, común en las vías de las lectinas y clásica). Este complejo es estabilizado por el factor P (properdina, que es sintetizada por neutrófilos activados por la presencia de patógenos y se almacena en gránulos secundarios).De este modo, se genera una C3 convertasa de membrana que genera un mayor número de moléculas C3b, gracias a la acción proteolítica del elemento Bb.
La C3 convertasa podrá unirse de nuevo a una molécula de C3b dando lugar a la C5 convertasa (C3b2Bb). Esta enzima, la C5 convertasa, tiene afinidad por la molécula de C5 y de nuevo es el elemento Bb el que presenta acción proteolítica, dando lugar a dos moléculas C5a y C5b. El elemento C5b será clave para la formación del complejo de ataque a la membrana (MAC), el cual se desarrollará más adelante.
Regulación de la vía alternativa:
El riguroso control al que está sujeta la activación del complemento explica cómo, en condiciones normales, C3b se pega solo en membranas de células patogénicas y no a nuestras propias células humanas.
La molécula de C3 está constituida por cadenas α y β unidas por un puente disulfuro, siendo la cadena α la de menor tamaño. Es en la cadena α dónde podemos encontrar un dominio especial que recibe el nombre de dominio TED (thioester-containing domain) qué contiene un enlace éster muy activo, formado por una cisteína y una glutamina entrelazadas por un átomo de azufre.
Normalmente C3 está plegado protegiendo el enlace tioéster, pero cuando actúa la convertasa, sobre C3 se libera C3a qué es el que protegía al enlace tioéster. C3b mantiene la estructura plegada, pero ahora con el dominio TED libre, que ahora puede interaccionar con grupos hidroxilo o amino que se encuentran en la superficie bacteriana en mayor proporción que en células humanas.
Si C3b no encuentra un sitio donde realizar la unión este se hidroliza rápidamente inactivándose. Así es como se inactiva la vía alternativa en individuos sanos.
En circunstancias normales o durante la activación del C’ puede pegarse C3b a células de organismo. Por ello, para regular el sistema del complemento existen proteínas reguladoras del complemento, que impiden que las células continúen con el proceso de activación del complemento. Está proteínas tienen como diana el elemento C3b. Son:
- CD55 o DAF (Decay Accelerating Factor)
- CD46 o MCP (Membrane Cofactor of Proteolysis)
- Proteína M
- Factor I
- CD35 o CR1 (Cell-surface complement Receptor type I)
Las tres primeras son proteínas específicas de especie, que impiden que el factor Bb se una a C3b. MCP actúa uniéndose a C3 convertasa, impidiendo la unión del factor P (properdina). Además, la proteína M y DAF actúan también compitiendo con Bb.
Además de evitar la unión de C3b al resto de elementos del complemento, es necesario que C3b se elimine para evitar la formación de la C3 convertasa. Esto se consigue gracias al factor I, que corta un pequeño fragmento de C3b y lo inactiva, dando lugar a C3f y iC3b. A continuación, iC3b es escindido por el factor I y por CR1, liberando C3c Y C3dg, qué queda unión a la membrana.
Vía de las lectinas
En las membranas existen estructuras formadas por diferentes glúcidos que distinguen las estructura de procariotas de la de eucariotas:
- Gram + : Ácido lipoteicoico
- Gram - : Lipopolisacáridos
- Hongos : Glicanos acabados en residuos de manosa
Los organismos vertebrados presentan una estructura modificada a lo largo de la evolución,
también con residuos de manosa pero en menor cantidad, y con otros residuos adicionales
como galactosa, fucosa y ácido siálico (N-acetilneuramínico).
Estos patrones que difieren entre los distintos organismos, son los que van a
ser reconocidos en la vía de las lectinas. En está vía ya si se distinguen proteínas de
reconocimiento especializadas.
En está vía distinguimos 2 receptores principales:
- MBL (mannose-binding lectin):
Se trata de una proteína de unión a manosa constituida por 6 módulos cada uno de los cuales consta de 3 monómeros, por lo que en total hay 18 moléculas formando una MLB. Estas proteínas son las que se van a encargar de reconocer los distintos patrones de distribución de la manosa en la superficie de patógenos.
Presenta una estructura similar al colágeno en su región aminoterminal, y una región móvil y flexible de α-hélices. En el extremo carboxilo presenta una región globular dónde reside el reconocimiento (C-type lectin domain), con un dominio de unión a azúcares o estructuras hidrocarbonadas.
- Ficolinas:
Son moléculas similares a MBL en forma y estructura, que van a reconocer azúcares acetilados. En el extremo carboxilo presentan un dominio de unión a azúcares del tipo “Fibrinogen-like domain”, en lugar del C-type domain que presentan las MBL.
Tanto las ficolinas como las MBL se asocian con α - serín proteasas llamadas MASP (MBL-associated serine proteases): MASP-1, MASP-2 y MASP-3, que se unen como zimógenos.
Cuando MBL se une a la superficie de un patógeno por reconocimiento de las estructuras antes mencionadas, ocurre un cambio conformacional en MASP-1 que le permite romper y activar a MASP-2, la cual, activada, podrá cortar componentes del sistema del complemento como C4 dando lugar a C4b y C4a. C4b queda adherido, mientras que C4a queda libre, con acción quimiotáctica.
A continuación, MASP-2 actúa sobre C2, produciendo C2b y C2a, siendo este último el fragmento de mayor tamaño y el que se unirá a C4b, formando la C3 convertasa de la vía de las lectinas (C4bC2a), siendo el elemento C2a el que presenta acción proteolítica.
Este complejo actúa sobre moléculas de C3 que escinde en C3b y C3a. Cuando una molécula de C3b se una al complejo C4bC2a formará el complejo C5 convertasa (C4bC2aC3b).
Es importante mencionar que toda la cascada proteolítica ocurre anclada a la membrana del patógeno.
Vía clásica
Esta vía es muy similar a la vía de las lectinas, solo que ahora en lugar de ser una ficolina o una MBL lo que reconozca patrones de la superficie de patógenos, el complejo molecular se llama C1q. Por tanto, prácticamente sólo varía el reconocimiento.
Ahora C1q detecta cambios conformacionales de la región constante de las inmunoglobulinas unidas a la bacteria. Además, también puede unirse a patrones específicos de la superficie del patógeno.
Cuando una bacteria entra en el organismo, hay anticuerpos naturales que la reconocen y se unen a ella. Estos anticuerpos sufren variaciones conformacionales que son detectadas por C1q, que activa proteínas del complemento.
C1q consta de dos moléculas duplicadas similares a MASP-1 y MASP-2 llamadas C1r y C1s (serín proteasas que inicialmente están en su forma inactiva). Además, C1q es un hexámero de trímeros compuestos por monómeros con un extremo aminoterminal globular y un extremo carboxiterminal con un dominio similar al del colágeno. Los trímeros se ensamblan a través de sus dominios carboxiterminales, dejando que los dominios globulares formen una estructura en forma de cabeza globular.
El reconocimiento reside en las 6 cabezas globulares de C1q. Cuando dos o más de estas cabezas interaccionan con su ligando, se produce un cambio conformacional en el complejo C1r:C1s que lleva a la activación de la actividad autocatalítica de C1r, que es capaz de romper y activar a C1s. C1s actúa sobre los componentes del sistema del complemento C4 y C2. La unión de C4b y C2a da lugar a la C3 convertasa, que al actuar sobre C3 genera C3b qué puede unirse a C4bC2a formando la C5 convertasa. Está actúan sobre moléculas de C5, produciendo C5a con acción quimiotáctica y C5b qué en fase soluble inicial el proceso de formación del complejo de ataque a la membrana.
NOTA: C4a actúa como un péptido mediador en la inflamación y C2b es un precursor de la quinina C2 vasoactiva. C3a y C5a actuarán también como mediadores en el proceso de inflamación.
Regulacion de la vía clásica
La unión del complejo C1s/C1r se inhibe por la acción de C1NH, que impide la unión con C1q. Así, se inactiva la vía clásica en su inicio, separando el complejo de proteínas de reconocimiento.
Existen enfermedades debidas a mutaciones del gen que codifica para C1NH, como el edema angioneurótico. En este caso, puede ocurrir que no haya C1NH o que haya cantidad suficiente, pero que carezca de funcionalidad.
Está enfermedad se caracteriza por la formación de edemas que ocluyen las vías respiratorias, y por un aumento en la permeabilidad vascular. Esto puede cursar con un edema intestinal que genera un dolor agudo, similar al de la peritonitis, confundiendo en el diagnóstico.
Formación del complejo de ataque a membrana (MAC)
El elemento C5b generado por la acción de la C5 convertasa por cualquiera de las anteriores vías no se pega a la membrana, sino que tiene afinidad por el C6 y juntos favorecen la unión con C7 dando lugar al complejo C7 (C5b-C6-C7).
Entonces el complejo se adhiere suavemente a la membrana, sin atravesarla, y queda anclado tras la llegada de C8. Posteriormente, C9 se une a C8 y se inicia el proceso de polimerización qué va a dar lugar a la formación de un poro en la membrana (10 nm de diámetro y 15 nm de altura), condicionando la lisis de la célula por choque osmótico.
Regulacion de la formación del MAC
La proteína CD59, también llamada protectina, se localiza en la membrana de las células
del hospedador, e impide el ensamblaje del complejo de ataque a nivel de C8 y C9.
CD59 y DAF presentan un grupo GPI (glicosilfosfatidilinositol) mediante el cual se unen a la
membrana codificado por el gen PIGA, en el cromosoma X.
Existe una enfermedad causada por el déficit de CD59 y DAF, llamada hemoglobinuria
paroxística nocturna, en la que la actividad normal del complemento no se detiene y se
lisan los hematíes, dando lugar a la aparición de hemoglobina en orina y a la excreción de
orina de color oscuro. Esto se debe a una mutación en el gen que regula la adecuada
síntesis de inositol, por lo que las proteínas reguladoras no se unen y no ejercen su
correcta función.
Trabajo realizado por Iman L.B., Marcos M.B., Mª Marina M.V. & Alberto Manuel P.P.
Sistema del Complemento (C')
Paralelamente a la barrera de los agentes antimicrobianos, entra en juego el sistema del complemento.
Consta de un gran número de proteínas plasmáticas solubles, localizadas en sangre u otros fluidos, que interactúan tanto para opsonizar patógenos como para inducir la respuesta inflamatoria contra la infección. Muchas de estas proteínas son proteasas que se activan tras su división por otras proteasas específicas.
Este sistema puede activarse por tres vías:
- Vía clásica: es la más reciente evolutivamente, y la primera en ser descubierta.
- Vía alternativa:es la más antigua evolutivamente, descrita tras la vía clásica.
- Vía de las lectinas:ocupa un lugar intermedio en la evolución, y es la más reciente en ser caracterizada.
Independientemente de la vía que se siga, la activación del complemento consta de una serie de fases:
- Fase de reconocimiento (3 vías): Es una fase muy delimitada en la vía de las lectinas y en la vía clásica. En la vía alternativa la situación es algo diferente ya que la activación se produce de forma intrínseca sin reconocimiento previo.
- Generación de la C3 y C5 convertasa: mediante una cascada enzimática mediada por zimógenos.
- Formación del complejo de ataque a la membrana: MAC (membrane attack complex) y la C5b forman un poro en la membrana que lisa la bacteria.
NOTA: A partir de ahora, se hará uso de una nomenclatura en la que el fragmento mayor va a asociado a la letra b (C4b, C3b y C5b) salvo en el caso de C2, en el que C2a es el fragmento mayor.
Vía alternativa
La característica principal de está vía es que de forma espontánea el factor C3, que presenta alta afinidad por el agua, se une a una molécula de agua, permitiendo la unión del factor B en fase soluble.
A continuación una proteasa especial de la vía, llamada factor D, se une al complejo C3(H2O)B y escinde al factor B en dos fragmentos liberándose el fragmento Ba y formándose la convertasa C3 soluble llamada C3(H2O)Bb.
Esta C3 convertasa soluble, una proteasa de residuos de serina, va a actuar sobre moléculas de C3, dando lugar a C3a y C3b. La acción proteolítica reside en el elemento Bb. El C3b quedará unido a la superficie bacteriana mediante un enlace tioéster (actuando a modo de marcador para la opsonización) mientras que el C3a liberado tendrá acción quimiotáctica.
C3b en la membrana se une de nuevo al factor B, que tras ser escindido por el factor D y liberar Ba, formará el C3bBb (C3 convertasa de membrana, común en las vías de las lectinas y clásica). Este complejo es estabilizado por el factor P (properdina, que es sintetizada por neutrófilos activados por la presencia de patógenos y se almacena en gránulos secundarios).De este modo, se genera una C3 convertasa de membrana que genera un mayor número de moléculas C3b, gracias a la acción proteolítica del elemento Bb.
La C3 convertasa podrá unirse de nuevo a una molécula de C3b dando lugar a la C5 convertasa (C3b2Bb). Esta enzima, la C5 convertasa, tiene afinidad por la molécula de C5 y de nuevo es el elemento Bb el que presenta acción proteolítica, dando lugar a dos moléculas C5a y C5b. El elemento C5b será clave para la formación del complejo de ataque a la membrana (MAC), el cual se desarrollará más adelante.
Regulación de la vía alternativa:
El riguroso control al que está sujeta la activación del complemento explica cómo, en condiciones normales, C3b se pega solo en membranas de células patogénicas y no a nuestras propias células humanas.
La molécula de C3 está constituida por cadenas α y β unidas por un puente disulfuro, siendo la cadena α la de menor tamaño. Es en la cadena α dónde podemos encontrar un dominio especial que recibe el nombre de dominio TED (thioester-containing domain) qué contiene un enlace éster muy activo, formado por una cisteína y una glutamina entrelazadas por un átomo de azufre.
Normalmente C3 está plegado protegiendo el enlace tioéster, pero cuando actúa la convertasa, sobre C3 se libera C3a qué es el que protegía al enlace tioéster. C3b mantiene la estructura plegada, pero ahora con el dominio TED libre, que ahora puede interaccionar con grupos hidroxilo o amino que se encuentran en la superficie bacteriana en mayor proporción que en células humanas.
Si C3b no encuentra un sitio donde realizar la unión este se hidroliza rápidamente inactivándose. Así es como se inactiva la vía alternativa en individuos sanos.
En circunstancias normales o durante la activación del C’ puede pegarse C3b a células de organismo. Por ello, para regular el sistema del complemento existen proteínas reguladoras del complemento, que impiden que las células continúen con el proceso de activación del complemento. Está proteínas tienen como diana el elemento C3b. Son:
- CD55 o DAF (Decay Accelerating Factor)
- CD46 o MCP (Membrane Cofactor of Proteolysis)
- Proteína M
- Factor I
- CD35 o CR1 (Cell-surface complement Receptor type I)
Las tres primeras son proteínas específicas de especie, que impiden que el factor Bb se una a C3b. MCP actúa uniéndose a C3 convertasa, impidiendo la unión del factor P (properdina). Además, la proteína M y DAF actúan también compitiendo con Bb.
Además de evitar la unión de C3b al resto de elementos del complemento, es necesario que C3b se elimine para evitar la formación de la C3 convertasa. Esto se consigue gracias al factor I, que corta un pequeño fragmento de C3b y lo inactiva, dando lugar a C3f y iC3b. A continuación, iC3b es escindido por el factor I y por CR1, liberando C3c Y C3dg, qué queda unión a la membrana.
Vía de las lectinas
En las membranas existen estructuras formadas por diferentes glúcidos que distinguen las estructura de procariotas de la de eucariotas:
- Gram + : Ácido lipoteicoico
- Gram - : Lipopolisacáridos
- Hongos : Glicanos acabados en residuos de manosa
Los organismos vertebrados presentan una estructura modificada a lo largo de la evolución, también con residuos de manosa pero en menor cantidad, y con otros residuos adicionales como galactosa, fucosa y ácido siálico (N-acetilneuramínico). Estos patrones que difieren entre los distintos organismos, son los que van a ser reconocidos en la vía de las lectinas. En está vía ya si se distinguen proteínas de reconocimiento especializadas.
En está vía distinguimos 2 receptores principales:
- MBL (mannose-binding lectin):
Se trata de una proteína de unión a manosa constituida por 6 módulos cada uno de los cuales consta de 3 monómeros, por lo que en total hay 18 moléculas formando una MLB. Estas proteínas son las que se van a encargar de reconocer los distintos patrones de distribución de la manosa en la superficie de patógenos. Presenta una estructura similar al colágeno en su región aminoterminal, y una región móvil y flexible de α-hélices. En el extremo carboxilo presenta una región globular dónde reside el reconocimiento (C-type lectin domain), con un dominio de unión a azúcares o estructuras hidrocarbonadas.
- Ficolinas:
Son moléculas similares a MBL en forma y estructura, que van a reconocer azúcares acetilados. En el extremo carboxilo presentan un dominio de unión a azúcares del tipo “Fibrinogen-like domain”, en lugar del C-type domain que presentan las MBL.
Tanto las ficolinas como las MBL se asocian con α - serín proteasas llamadas MASP (MBL-associated serine proteases): MASP-1, MASP-2 y MASP-3, que se unen como zimógenos.
Cuando MBL se une a la superficie de un patógeno por reconocimiento de las estructuras antes mencionadas, ocurre un cambio conformacional en MASP-1 que le permite romper y activar a MASP-2, la cual, activada, podrá cortar componentes del sistema del complemento como C4 dando lugar a C4b y C4a. C4b queda adherido, mientras que C4a queda libre, con acción quimiotáctica.
A continuación, MASP-2 actúa sobre C2, produciendo C2b y C2a, siendo este último el fragmento de mayor tamaño y el que se unirá a C4b, formando la C3 convertasa de la vía de las lectinas (C4bC2a), siendo el elemento C2a el que presenta acción proteolítica.
Este complejo actúa sobre moléculas de C3 que escinde en C3b y C3a. Cuando una molécula de C3b se una al complejo C4bC2a formará el complejo C5 convertasa (C4bC2aC3b).
Es importante mencionar que toda la cascada proteolítica ocurre anclada a la membrana del patógeno.
Vía clásica
Esta vía es muy similar a la vía de las lectinas, solo que ahora en lugar de ser una ficolina o una MBL lo que reconozca patrones de la superficie de patógenos, el complejo molecular se llama C1q. Por tanto, prácticamente sólo varía el reconocimiento.
Ahora C1q detecta cambios conformacionales de la región constante de las inmunoglobulinas unidas a la bacteria. Además, también puede unirse a patrones específicos de la superficie del patógeno.
Cuando una bacteria entra en el organismo, hay anticuerpos naturales que la reconocen y se unen a ella. Estos anticuerpos sufren variaciones conformacionales que son detectadas por C1q, que activa proteínas del complemento.
C1q consta de dos moléculas duplicadas similares a MASP-1 y MASP-2 llamadas C1r y C1s (serín proteasas que inicialmente están en su forma inactiva). Además, C1q es un hexámero de trímeros compuestos por monómeros con un extremo aminoterminal globular y un extremo carboxiterminal con un dominio similar al del colágeno. Los trímeros se ensamblan a través de sus dominios carboxiterminales, dejando que los dominios globulares formen una estructura en forma de cabeza globular.
El reconocimiento reside en las 6 cabezas globulares de C1q. Cuando dos o más de estas cabezas interaccionan con su ligando, se produce un cambio conformacional en el complejo C1r:C1s que lleva a la activación de la actividad autocatalítica de C1r, que es capaz de romper y activar a C1s. C1s actúa sobre los componentes del sistema del complemento C4 y C2. La unión de C4b y C2a da lugar a la C3 convertasa, que al actuar sobre C3 genera C3b qué puede unirse a C4bC2a formando la C5 convertasa. Está actúan sobre moléculas de C5, produciendo C5a con acción quimiotáctica y C5b qué en fase soluble inicial el proceso de formación del complejo de ataque a la membrana.
Regulacion de la vía clásica
La unión del complejo C1s/C1r se inhibe por la acción de C1NH, que impide la unión con C1q. Así, se inactiva la vía clásica en su inicio, separando el complejo de proteínas de reconocimiento.Existen enfermedades debidas a mutaciones del gen que codifica para C1NH, como el edema angioneurótico. En este caso, puede ocurrir que no haya C1NH o que haya cantidad suficiente, pero que carezca de funcionalidad.
Está enfermedad se caracteriza por la formación de edemas que ocluyen las vías respiratorias, y por un aumento en la permeabilidad vascular. Esto puede cursar con un edema intestinal que genera un dolor agudo, similar al de la peritonitis, confundiendo en el diagnóstico.
Formación del complejo de ataque a membrana (MAC)
El elemento C5b generado por la acción de la C5 convertasa por cualquiera de las anteriores vías no se pega a la membrana, sino que tiene afinidad por el C6 y juntos favorecen la unión con C7 dando lugar al complejo C7 (C5b-C6-C7).
Entonces el complejo se adhiere suavemente a la membrana, sin atravesarla, y queda anclado tras la llegada de C8. Posteriormente, C9 se une a C8 y se inicia el proceso de polimerización qué va a dar lugar a la formación de un poro en la membrana (10 nm de diámetro y 15 nm de altura), condicionando la lisis de la célula por choque osmótico.
Regulacion de la formación del MAC
La proteína CD59, también llamada protectina, se localiza en la membrana de las células
del hospedador, e impide el ensamblaje del complejo de ataque a nivel de C8 y C9.
CD59 y DAF presentan un grupo GPI (glicosilfosfatidilinositol) mediante el cual se unen a la
membrana codificado por el gen PIGA, en el cromosoma X.
Existe una enfermedad causada por el déficit de CD59 y DAF, llamada hemoglobinuria
paroxística nocturna, en la que la actividad normal del complemento no se detiene y se
lisan los hematíes, dando lugar a la aparición de hemoglobina en orina y a la excreción de
orina de color oscuro. Esto se debe a una mutación en el gen que regula la adecuada
síntesis de inositol, por lo que las proteínas reguladoras no se unen y no ejercen su
correcta función.

Trabajo realizado por Iman L.B., Marcos M.B., Mª Marina M.V. & Alberto Manuel P.P.
