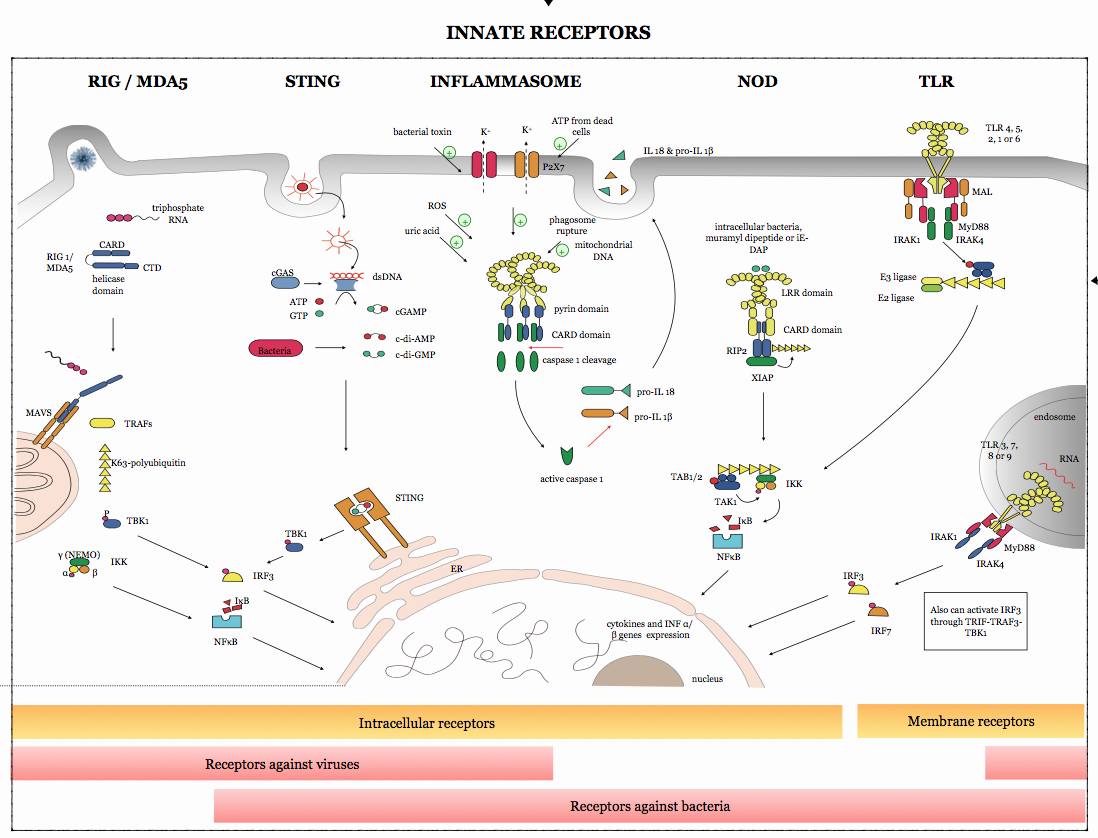
TLR (Toll Like Receptor)
El biólogo Jules Hoffmann descubrió los receptores Toll en D.melanogaster mientras intentaba demostrar el mecanismo de transducción en insectos de un factor nuclear denominado NF𝜅B.
Hoffmann descubrió que el gen Toll regulaba la expresión de NF𝜅B, y tenía una doble misión:
- En primer lugar, estaba relacionado con la diferenciación dorso-ventral de la mosca.
- Ejercía un papel importante en la defensa frente a hongos, mediante la producción de agentes antimicrobianos como la drosomicina.
En organismos distintos a D.melanogaster, encontramos análogos a los Toll en animales denominados TLR (Toll-like receptors), que participan en la defensa de no sólo hongos, sino también frente a virus y bacterias.
Estos TLR ejercen su función reconociendo lo que se conoce como PAMPs (Pathogen-associated molecular patterns), como el ácido lipoteicoico en Gram + y el LPS en Gram -, además de flagelos con subunidades de flagelina y elementos CpG no metilados.
Estos TLR presentan una especificidad limitada en comparación con los receptores de la inmunidad adaptativa. Sin embargo, pueden reconocer un amplio rango de elementos propios de microorganismos patógenos, y se expresan en muchos tipos celulares, como macrófagos o células dendríticas.
- Estructura de los TLR.
Son proteínas transmembrana con una región extracelular formada por 18-25 copias de repeticiones ricas en leucina, LRR (Leucine-rich repeat). Cada repetición consta de 20-25 aminoácidos, y múltiples LRRs forman una estructura con una morfología similar a la de una herradura que reconoce y une ligandos.
Presentan 3 dominios:
- - Dominio extracelular, con LRRs.
- Región convexa. Interacciona con el ligando. Presenta regiones con α-hélices (que aportan flexibilidad) y giros.
- Región cóncava. Tiene las repeticiones de leucina LRR (zonas de bandas de hebras β)
- - Dominio transmembrana.
- - Dominio intracitoplasmático, con un dominio TIR (Toll-IL-1 receptor) que inicia todo el proceso de transducción de señal tras la unión del ligando.
- Los TLR se pueden localizar en :
- - Membrana plasmática, al igual que los receptores Toll de D. melanogaster.
- - Membrana del endosoma, desde dónde detectan patógenos , o productos patológicos, introducidos a la célula por procesos de fagocitosis, macropinocitosis o endocitosis mediada por receptores.
- Distribución de los TLR
- TLRs en membrana plasmática (macrófagos y células dendríticas)
- Homodímeros
- - TLR-4: reconoce lipopolisacáridos característicos de las bacterias Gram - y ácidos lipoteicoicos de bacterias Gram+
- - TLR-5: reconoce la flagelina de los flagelos bacterianos. Reconoce un dominio altamente conservado y escondido, por lo que sólo se une a flagelina monomérica de bacterias descompuestas en el espacio extracelular.
- Heterodímeros
- - TLR-2 + TLR-6: reconoce lipopéptidos con 2 cadenas de ácidos grasos (diacil lipopéptidos)
- - TLR-2 + TLR-1: reconoce lipopéptidos con 3 cadenas de ácidos grasos (triacil lipopéptidos)
- TLRs en endosomas
Son homodímeros. Son TLRs más hidrofílicos ya que reconocen ácidos nucleicos.
- Reconocimiento de estructura de RNA
- - TLR-3: reconoce RNA de doble cadena que se internaliza por endocitosis directa de virus o fagocitosis de células infectadas moribundas. Se localiza en macrofagos, células dendriticas convencionales y células epiteliales intestinales.
- - TLR-7 y TLR-8. RNA de cadena simple. Característico de ortovirus (influenza, flavivirus…). Los TLR-7 se localizan en células dendríticas plasmocitoides y eosinófilos, mientras que los TLR-8 lo hacen en monocitos y macrófagos. Polimorfismos en el gen de TLR-7 están asociados a un mayor riesgo de lupus eritematoso sistémico.
- Reconocimiento de estructura de DNA
- - TLR9: reconoce estructuras de DNA tanto virales como bacterianos. Reconoce los islotes CpG no metilados (en eucariotas normalmente están metilados por la acción de una DNA metiltransferasa). Se localizan en células dendríticas plasmocitoides y en eosinófilos.
- Vías de transducción de señal de los TLR.
- TLR-4
No todos los TLR se unen directamente a sus ligandos. Esto se pone de manifiesto en el caso de TLR-4, que emplea dos proteínas accesorias para el reconocimiento del lipopolisacárido:
- MD-2: se une a TLR4 en la célula, y se necesita para, además del reconocimiento del LPS, para el correcto tráfico de TLR-4 a la superficie bacteriana.
MD-2 se asocia a la región central del dominio en forma de herradura de TLR-4. Cuando este complejo se une al LPS, cinco cadenas se unen al bolsillo hidrófobo de MD-2, pero no directamente a TLR-4. La sexta cadena permanece expuesta en la superficie de MD-2, y se une a la zona convexa de un segundo TLR-4, induciendo la dimerización.
- LBP: se une al LPS en fase soluble y se lo transfiere a CD14, que se encuentra localizado en la superficie de macrofagos, células dendriticas y neutrofilos.
- Es importante que no haya LPS libre en sangre, porque puede causar un colapso del sistema circulatorio o shock séptico, ya que el LPS induce la producción descontrolada de TNF-α, causando una permeabilidad vascular sistémica, disminuyendo drásticamente la presión arterial, lo que culmina en paro cardíaco.
La unión del ligando causa un cambio conformacional en el dominio TIR del receptor. MyD88 (Myeloid differentiation primary response 88) (o también MAL((MyD88-adapter-like), que es otra molécula adaptadora) también tiene un dominio TIR en su extremo carboxilo que se asocia con el dominio TIR del receptor.
En su extremo amino, MyD88 tiene un dominio de muerte DD (death domain) que se asocia con otros dominios DD de otras proteínas intracelulares señalizadoras.
El dominio DD de MyD88 recluta y activa dos serin-treonin protein quinasas, primero IRAK-1 y luego IRAK-4, activada por IRAK-1 (interleukin receptor activated kinase) a través de sus respectivos dominios DD.
Estas quinasas van a participar en la formación de un andamio o “scaffold” mediante el reclutamiento de TRAF6 (TNF receptor associated factor), una E3 ubiquitin ligasa, que actúa cooperativamente con UBC13 (Ubiquitin-conjugating enzyme E2), una E2 ubiquitin ligasa, y su cofactor Uve1A. UBC13 y Uve1A reciben el nombre de TRIKA.
Estas moléculas ubiquitinizan en la lisina 63 formando enlaces K63, dando lugar a polímeros de ubiquitina que actúan como andamios o plataformas de anclaje para la unión de otras proteínas.
Este andamio va a reclutar a TAB1 y TAB2 (TAK1-binding protein 1) y a la serin-treonin quinasa TAK1 (TGF-beta activated kinase 1). A continuación, TAK1 es fosforilada por IRAK-4 quedando activada y propagando la señal mediante la activación de una cascada de MAPKs, como JNK (C-Jun terminal kinase), que inducen la transcripción de genes de citoquinas.
Además, TAK1 fosforila y activa al complejo kinasas IkB (IKK) constituido por IKKα, IKKβ e IKKγ (también conocido como NEMO). NEMO se une a la cadena de poliubiquitina y aproxima todo el complejo IKK a TAK1, que fosforila y activa a IKKβ. Está a su vez fosforila a IkB (inhibidor de NFkB, constituido por dos subunidades p50 y p65). La molécula de IkB se degrada y libera a NFkB, que se transloca al núcleo donde actúa favoreciendo la expresión de genes de citoquinas proinflamatorias IL-1β, TNF-α e IL-6, junto a quimiocinas IL-8 e IL-12. Esta actúa sobre los linfocitos CD4 TH para inducir su diferenciación a TH1 que potencian la respuesta celular de los propios macrófagos, NK y linfocitos CD8 citotóxicos.
- TLR-3
Utiliza de forma exclusiva la molécula adaptadora TRIF (TIR domain Receptor Induce Interferon β), con la que interacciona a través de su dominio TIR intracitoplasmático. TRIF recluta a TRAF3, una E3 ubiquitin ligasa, que junto con TRAF6 forman un andamio de poliubiquitina que servirá de anclaje a NEMO, TANK, IKKε y TBK1, favoreciendo la actividad quinasa de TBK1 que fosforilará a IRF3.
Finalmente, IRF3 se transloca al núcleo y actuará como factor de transcripción de los genes de interferón tipo I.
- TLR-7 y TLR-9
El proceso es similar al del TLR4.
El dominio TIR de MyD88 recluta al complejo IRAK1/IRAK4. IRAK1 puede asociarse fácilmente a IRF7, lo que hace que este quede fosforilado por la acción de IRAK1. Ahora IRAK1 actuará como factor de transcripción en el núcleo expresando los genes de interferón tipo I.
RIG-1 y MDA5
Cuando un virus intracelular penetra en la célula, este va a ser reconocido por una familia de proteínas denominadas receptores tipo RIG-I (RLR). Estos receptores se encuentran en el citoplasma de las células y no se encuentran anclados a la membrana. Su mecanismo de acción se basa en el reconocimiento de los virus RNA.
RIG-1 es capaz de discriminar entre el RNA viral y el propio, en función de las modificaciones postranscripcionales que presente dicho RNA.
Las tres posibles modificaciones post-transcripcionales son:
- RNA con un extremo trifosfato sin grupo 7 metil-guanosina en 5’
- Cap0: Este tipo de RNA va a tener un grupo 7 metil-guanosina en el extremo 5’
- Cap1: El RNA va a tener un grupo metilo en oxígeno 3’ del anillo de ribosa del primer nucleótido en 5’
- Cap2: El RNA va a tener un grupo metilo en el oxígeno 3’ del anillo de ribosa tanto en el primer como en el segundo nucleótido del extremo 5’.
Dado que la mayoría de los virus no se replican en el núcleo, qué es dónde ocurren las modificaciones que si afectan al RNA propio, RIG-1 podrá detectar los extremos 5’ trifosfato no metilados de los virus.
En el caso de los picornavirus, que carecen de extremo 5’ trifosfato, RIG-1 no es capaz de reconocerlos.
Estructura de RIG-I
Presenta dos dominios CARD (caspase recruitment domain) que interaccionan con moléculas adaptadoras para la inducir la producción de interferon tipo I, así como dominios similares a helicasa mediante los cuales se une a las moléculas de RNA.
Mecanismo de acción
En un principio, RIG-I se va a encontrar autoinhibido por el plegamiento de su secuencia aminoacídica y estabilizado por interacciones entre los dominios CARD y helicasa.. El reconocimiento del RNA sin 7-metil-guanosina en 5’ o los RNA cap0 y cap1 gracias a la histidina 830 de su centro catalítico, va a provocar un cambio conformacional, alterando las interacciones que estabilizaban el plegamiento y haciendo qué RIG-1 se active y se despliegue.
Este proceso, va a dejar expuesto unos dominios CARD de RIG-I que les van a permitir interaccionar con los dominios CARD de unas proteínas mitocondriales llamadas MAVS. La interacción con MAVS va a mediar el reclutamiento de TRAFs que van a polimerizar una cadena de ubiquitina, que servirá como andamiaje a las IKKs, las cuales, una vez activadas, fosforilarán a:
- - IRF-3: que se dirigirá al núcleo y mediará la expresión de genes de interferón tipo
- - IkB: que será degradado vía proteasoma y permitirá que NFkB se transloque al núcleo y favorecerá la expresión de citoquinas proinflamatorias.
NOTA: Junto con RIG-I existen otras moléculas que realizan funciones similares como son:
- - MAD-5 (asociado a la diferenciación de melanoma tipo 5): también llamado helicard, es similar en estructura a RIG-I, pero detecta dsRNA (como el de los Picornavirus)
- - LGP2 (de la familia de RLR (RIG-like receptors)) contiene un dominio helicasa pero no CARD y se encarga de cooperar con RIG-I y MAD-5.
STING
STING (Stimulator of interferon genes) es una proteína anclada al retículo endoplasmático, orientada hacia el citosol. Esta estructura se va a encargar de reconocer moléculas procedentes tanto de virus como de bacterias. Su acción está mediada por la proteína cGAS, ya que reconoce dinucleótidos cíclicos (CDNs) como c-di-GMP o c-di-AMP que actúan como segundos mensajeros producidos por bacterias y virus:
- Virus: Los virus DNA secretan su material genético en la célula y este activa a la proteína cGAS (Cyclic GMP-AMP (cGAMP) synthase) (que se une a ellos a través de estructuras denominadas dedos de Zinc). cGAS activa se va a encargar de la formación de heterodinucleótidos de guanina y adenina que van a ser reconocidos por STING.
- Bacterias: Las bacterias van a secretar homodinucleótidos de guanina y adenina que van a ser también reconocidos por STING.
En ambos casos, el reconocimiento de los dinucleótidos por STING va a provocar la dimerización de la proteína y la activación de TBK1, que va a ser fosforilada. Trás esto, TBK1 fosforilará a IRF3 que se translocará al núcleo favoreciendo la expresión de genes de interferón tipo I.
NOD
Mientras que los TLRs son principalmente elementos sensores de productos microbianos extracelulares, los NOD (nucleotide-binding oligomerization domain) son receptores citoplasmáticos sensores de productos microbianos que se van a encontrar en el citoplasma.
Los NOD presentan un dominio de oligomerización de unión a nucleótidos, que va a ser el que favorezca la activación del mismo y la consecuente respuesta celular. Se han descubierto otras familias similares a NOD conocidas como NLR (NOD like receptors) que también intervienen en proceso de la respuesta innata.
Estructura de los NOD.
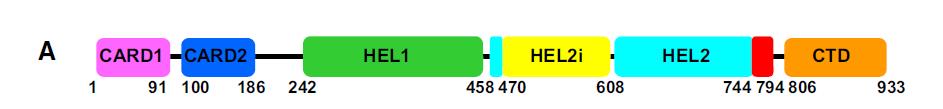
La estructura de los NOD va a estar constituida por dos dominios CARDs, el dominio NBD y un dominio LRR similar al de los TLRs. El dominio LRR es el que les va a permitir sensar esos productos microbianos creados por la bacteria.
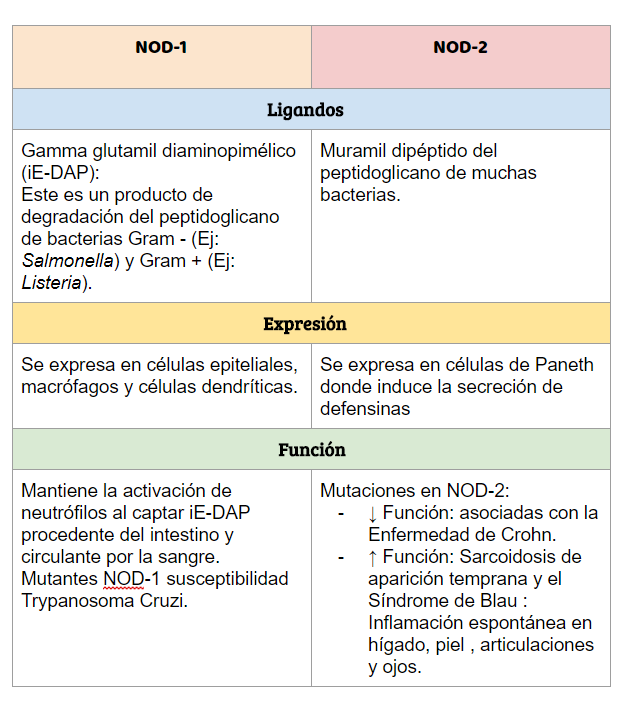
A continuación recogemos una tabla resumen de los NOD más estudiados con sus ligandos:
Mecanismo de acción:
Una vez activado NOD por uno de sus ligandos antes mencionados en la tabla, se va a producir la dimerización de la proteína y el reclutamiento de RIP2 (Receptor-interacting serine/threonine-protein kinase 2) y XIAP (X-linked inhibitor of apoptosis protein) gracias a sus dominios CARD.
XIAP va a generar un andamio de poliubiquitina qué va a servir de estructura de anclaje para TAB1/2, TAK1 y NEMO junto con IKKα e IKKβ. TAK1 se activará por fosforilación, y tras esto fosforilará a IKKβ.
IKKβ favorecerá la degradación de IkB mediante su fosforilación, y permitirá la liberación de NFkB que se translocará al núcleo y favorecerá la expresión de citoquinas proinflamatorias.
Inflamasoma
El inflamasoma es un conjunto de moléculas que inducen la producción de citocinas proinflamatorias :IL-1β ,IL-18 e IL-33.
Constituidas por:
- - Una molécula sensora (NLR)
- - Otra adaptadora (ASC)
- - Otra efectora (caspasa 1).
La caspasa 1 requiere un procesamiento previo para actuar que se realiza en el inflamasoma. Su acción es escindir los propéptido de las citocinas proinflamatorias para generar las formas activas que se liberan al medio para ejercer su función. En este grupo se incluyen aquellas moléculas que presentan un dominio pirina, y se les conoce como la familia de los NLRP. A continuación veremos cuatro de las familias más estudiadas de los NLRP:
NLRP3
Reside de forma inactiva en el citoplasma, donde sus dominios LRR se unen a la chaperona HSP90 y a la co-chaperona SGT1.
Existen unos determinados eventos que inducen la señalización por NLRP3:
- disminución de K+ intracelular → causa la disociación de HSP90 y SGT1.
- generación de ROS
- disrupción de lisosomas.
La disminución de K+ se da durante la infección debido a la formación de poros por toxinas de microorganismos patógenos como S.aureus.
El modelo de señalización de NLRP3 mediado por ROS implica la oxidación de proteínas sensoras TRX (tiorredoxinas) que normalmente están unidas a TXNIP (thioredoxin-interacting protein) pero que al oxidarse se disocian, causando que la TXNIP libre desplace a la HSP90 y a SGT1, induciendo la activación.
Por último, la fagocitosis de sustancias, como el adyuvante alum, una sal cristalina de aluminio, potasio y sulfatos, puede provocar la ruptura de lisosomas y la liberación de catepsina B activa, que estimula la vía NLRP3.
Mecanismo de acción:
NLRP3 induce la producción de citoquinas pro-inflamatorias y la muerte celular mediante un complejo multiproteico, el inflamasoma, que actúa en varias etapas.
El proceso se inicia con la agregación de dominios LRR de varias moléculas de NLRP3 en respuesta a un reconocimiento desencadenante. La agregación induce la interacción de dominios pirina con los dominios pirina de la proteína ASC (apoptosis-associated speck-like protein containing a carboxy-terminal CARD), una proteína adaptadora con un dominio pirina en el extremo aminoterminal y un dominio CARD en el extremo carboxiterminal.
La interacción causa la formación de un filamento ASC polimérico, que presenta el dominio pirina en el centro y los dominios CARD orientados hacia el exterior. Estos dominios CARD interaccionan con los dominios CARD de una proteasa inactiva, la pro-caspasa 1, iniciando un proceso de polimerización dependiente de CARD que da lugar a filamentos de caspasa 1
Está agregación desencadena el autoprocesamiento de la pro-caspasa 1, que libera el fragmento activo de caspasa 1, siendo ésta quien lleve a cabo el procesamiento proteolítico dependiente de ATP de citoquinas pro-inflamatorias, principalmente IL-1ꞵ y IL-18, en sus formas activas
La activación de caspasa 1 también induce una forma de muerte celular conocida como piroptosis, mediante un mecanismo desconocido aún.
NLRP1
Se da en monocitos y células dendríticas.
Se activa por los productos de B. anthracis que va a secretar dos proteínas: factor letal (LF) y antígeno protectivo (PA). PA genera un poro en la membrana del hospedador que usa a LF para entrar en la célula.
- - Si hay altas dosis de LF se activará NLRP1 y se producirá la muerte por apoptosis.
- - Si las dosis son bajas, NLRP1 se activa y se genera IL-1β.
Existen dos teorías sobre la activación de NLRP1:
- - Activación directa: LF corta un fragmento aminoterminal y se activa.
- - Activación indirecta: Existe una proteína desconocida que inhibe NLRP1. Entonces LF actuaría escindiendo a la proteína desconocida y activando el dominio CARD.
NLRC4
NLRC4 se incluye en ocasiones dentro del grupo de molécula adaptadoras, porque actúa como adaptador con otras dos proteínas NLR: NAIP2 y NAIP5 (NLR Family Apoptosis Inhibitory Protein), qué detectan proteínas bacterianas que entran en la célula mediante sistemas de secreción usados por patógenos para acceder a nutrientes o transportar materiales desde la célula hospedadora.
La proteína PrgJ es componente del sistema de secreción tipo 3 (T3SS), un complejo macromolecular con forma de aguja.
Cuando se produce la infección por patógenos como Salmonella , PrgJ entra a la célula y es reconocida por NLRC4 y por NAIP2. La flagelina extracelular es reconocida por TLR5, pero dado que no entra con PrgJ por T3SS, es reconocida por NLRC4 en conjunción con NAIP5.
El flagelo y T3SS tienen una estructura similar: en la base la estructura es idéntica, y la zona de transición es similar, pero en el caso de T3SS el interior está formado por múltiples cadenas de proteína Rod, y acaba finalmente en la región aguja, qué es lo que realmente penetra en el citoplasma celular.
El sistema de inyección inyecta flagelina, proteína Rod y PrgJ, así como monómeros de la propia proteína de inyección, PrgI.
Estas proteínas serán las qué activen el sistema del inflamasoma.
En bacterias como Shigella, que carecen de flagelos, existe un plásmido que codifica para el sistema de inyección. En este caso, sólo se produce Rod y proteína aguja.
Al inyectar las proteínas, se adhieren cada una a su correspondiente proteína NAIP y reclutan a ASC mediante la formación de una estructura en forma de roseta, con dos anillos, uno interno y otro externo. Está estructura es similar a la de los TLR, con regiones ricas en leucina.
AIM
La familia AIM2 (absent in melanoma 2)
es una familia incluida dentro de
los inflamasomas que se activa
por bacterias citosólicas y virus de
DNA. Se caracteriza por contener
el dominio HIN, que reconoce el DNA
de cadena doble y desencadena
la activación de la caspasa 1.
AIM2 se localiza en el citoplasma.
In vitro, se ha visto qué es importante
en la respuesta frente a Vaccinia virus (VACV). In vivo, su deficiencia
ha demostrado estar relacionada con
una mayor susceptibilidad a
infecciones por F. novicida.
Tiene dos vías de activación:
- - Vía no canónica de activación: esta vía se da cuando las bacterias son fagocitadas, como en el caso de Francisella novicida, y escapan de las vacuolas, favoreciendo la producción y activación de cGAS, la activación de STING e IFI204 e inducen la producción de IFN tipo I. Este IFN tipo I, a través de unos receptores de INF tipo I, median la inducción de GBPs los cuales actúan sobre la vacuola y, después, sobre el propio contenido de la bacteria. Esto genera la liberación del DNA bacteriano y la activación de AIM2.
- - Vía canónica de activación: se da en el caso de virus (como MCMV) o de DNA transfectado. Este DNA es reconocido por AIM2 activándose.
AIM2 activo en ambos casos, recluta a ASC como molécula mediadora y activa a la caspasa 1 provocando o la piroptosis celular o bien la activación de la pro-IL-1β a IL-1β
Trabajo realizado por Iman L.B., Marcos M.B., Mª Marina M.V. & Alberto Manuel P.P.
TLR (Toll Like Receptor)
El biólogo Jules Hoffmann descubrió los receptores Toll en D.melanogaster mientras intentaba demostrar el mecanismo de transducción en insectos de un factor nuclear denominado NF𝜅B.
Hoffmann descubrió que el gen Toll regulaba la expresión de NF𝜅B, y tenía una doble misión:
- En primer lugar, estaba relacionado con la diferenciación dorso-ventral de la mosca.
- Ejercía un papel importante en la defensa frente a hongos, mediante la producción de agentes antimicrobianos como la drosomicina.
En organismos distintos a D.melanogaster, encontramos análogos a los Toll en animales denominados TLR (Toll-like receptors), que participan en la defensa de no sólo hongos, sino también frente a virus y bacterias.
Estos TLR ejercen su función reconociendo lo que se conoce como PAMPs (Pathogen-associated molecular patterns), como el ácido lipoteicoico en Gram + y el LPS en Gram -, además de flagelos con subunidades de flagelina y elementos CpG no metilados.
Estos TLR presentan una especificidad limitada en comparación con los receptores de la inmunidad adaptativa. Sin embargo, pueden reconocer un amplio rango de elementos propios de microorganismos patógenos, y se expresan en muchos tipos celulares, como macrófagos o células dendríticas.
- Estructura de los TLR.
- - Dominio extracelular, con LRRs.
- Región convexa. Interacciona con el ligando. Presenta regiones con α-hélices (que aportan flexibilidad) y giros.
- Región cóncava. Tiene las repeticiones de leucina LRR (zonas de bandas de hebras β)
- - Dominio transmembrana.
- - Dominio intracitoplasmático, con un dominio TIR (Toll-IL-1 receptor) que inicia todo el proceso de transducción de señal tras la unión del ligando.
- Los TLR se pueden localizar en :
- - Membrana plasmática, al igual que los receptores Toll de D. melanogaster.
- - Membrana del endosoma, desde dónde detectan patógenos , o productos patológicos, introducidos a la célula por procesos de fagocitosis, macropinocitosis o endocitosis mediada por receptores.
Son proteínas transmembrana con una región extracelular formada por 18-25 copias de repeticiones ricas en leucina, LRR (Leucine-rich repeat). Cada repetición consta de 20-25 aminoácidos, y múltiples LRRs forman una estructura con una morfología similar a la de una herradura que reconoce y une ligandos.
Presentan 3 dominios:
- Distribución de los TLR
- TLRs en membrana plasmática (macrófagos y células dendríticas)
- Homodímeros
- - TLR-4: reconoce lipopolisacáridos característicos de las bacterias Gram - y ácidos lipoteicoicos de bacterias Gram+
- - TLR-5: reconoce la flagelina de los flagelos bacterianos. Reconoce un dominio altamente conservado y escondido, por lo que sólo se une a flagelina monomérica de bacterias descompuestas en el espacio extracelular.
- Heterodímeros
- - TLR-2 + TLR-6: reconoce lipopéptidos con 2 cadenas de ácidos grasos (diacil lipopéptidos)
- - TLR-2 + TLR-1: reconoce lipopéptidos con 3 cadenas de ácidos grasos (triacil lipopéptidos)
- TLRs en endosomas
- Reconocimiento de estructura de RNA
- - TLR-3: reconoce RNA de doble cadena que se internaliza por endocitosis directa de virus o fagocitosis de células infectadas moribundas. Se localiza en macrofagos, células dendriticas convencionales y células epiteliales intestinales.
- - TLR-7 y TLR-8. RNA de cadena simple. Característico de ortovirus (influenza, flavivirus…). Los TLR-7 se localizan en células dendríticas plasmocitoides y eosinófilos, mientras que los TLR-8 lo hacen en monocitos y macrófagos. Polimorfismos en el gen de TLR-7 están asociados a un mayor riesgo de lupus eritematoso sistémico.
- Reconocimiento de estructura de DNA
- - TLR9: reconoce estructuras de DNA tanto virales como bacterianos. Reconoce los islotes CpG no metilados (en eucariotas normalmente están metilados por la acción de una DNA metiltransferasa). Se localizan en células dendríticas plasmocitoides y en eosinófilos.
- Vías de transducción de señal de los TLR.
- TLR-4
No todos los TLR se unen directamente a sus ligandos. Esto se pone de manifiesto en el caso de TLR-4, que emplea dos proteínas accesorias para el reconocimiento del lipopolisacárido:
- MD-2: se une a TLR4 en la célula, y se necesita para, además del reconocimiento del LPS, para el correcto tráfico de TLR-4 a la superficie bacteriana. MD-2 se asocia a la región central del dominio en forma de herradura de TLR-4. Cuando este complejo se une al LPS, cinco cadenas se unen al bolsillo hidrófobo de MD-2, pero no directamente a TLR-4. La sexta cadena permanece expuesta en la superficie de MD-2, y se une a la zona convexa de un segundo TLR-4, induciendo la dimerización.
- LBP: se une al LPS en fase soluble y se lo transfiere a CD14, que se encuentra localizado en la superficie de macrofagos, células dendriticas y neutrofilos.
- Es importante que no haya LPS libre en sangre, porque puede causar un colapso del sistema circulatorio o shock séptico, ya que el LPS induce la producción descontrolada de TNF-α, causando una permeabilidad vascular sistémica, disminuyendo drásticamente la presión arterial, lo que culmina en paro cardíaco.
La unión del ligando causa un cambio conformacional en el dominio TIR del receptor. MyD88 (Myeloid differentiation primary response 88) (o también MAL((MyD88-adapter-like), que es otra molécula adaptadora) también tiene un dominio TIR en su extremo carboxilo que se asocia con el dominio TIR del receptor.
En su extremo amino, MyD88 tiene un dominio de muerte DD (death domain) que se asocia con otros dominios DD de otras proteínas intracelulares señalizadoras.
El dominio DD de MyD88 recluta y activa dos serin-treonin protein quinasas, primero IRAK-1 y luego IRAK-4, activada por IRAK-1 (interleukin receptor activated kinase) a través de sus respectivos dominios DD.
Estas quinasas van a participar en la formación de un andamio o “scaffold” mediante el reclutamiento de TRAF6 (TNF receptor associated factor), una E3 ubiquitin ligasa, que actúa cooperativamente con UBC13 (Ubiquitin-conjugating enzyme E2), una E2 ubiquitin ligasa, y su cofactor Uve1A. UBC13 y Uve1A reciben el nombre de TRIKA.
Estas moléculas ubiquitinizan en la lisina 63 formando enlaces K63, dando lugar a polímeros de ubiquitina que actúan como andamios o plataformas de anclaje para la unión de otras proteínas.
Este andamio va a reclutar a TAB1 y TAB2 (TAK1-binding protein 1) y a la serin-treonin quinasa TAK1 (TGF-beta activated kinase 1). A continuación, TAK1 es fosforilada por IRAK-4 quedando activada y propagando la señal mediante la activación de una cascada de MAPKs, como JNK (C-Jun terminal kinase), que inducen la transcripción de genes de citoquinas.
Además, TAK1 fosforila y activa al complejo kinasas IkB (IKK) constituido por IKKα, IKKβ e IKKγ (también conocido como NEMO). NEMO se une a la cadena de poliubiquitina y aproxima todo el complejo IKK a TAK1, que fosforila y activa a IKKβ. Está a su vez fosforila a IkB (inhibidor de NFkB, constituido por dos subunidades p50 y p65). La molécula de IkB se degrada y libera a NFkB, que se transloca al núcleo donde actúa favoreciendo la expresión de genes de citoquinas proinflamatorias IL-1β, TNF-α e IL-6, junto a quimiocinas IL-8 e IL-12. Esta actúa sobre los linfocitos CD4 TH para inducir su diferenciación a TH1 que potencian la respuesta celular de los propios macrófagos, NK y linfocitos CD8 citotóxicos. - TLR-3
Utiliza de forma exclusiva la molécula adaptadora TRIF (TIR domain Receptor Induce Interferon β), con la que interacciona a través de su dominio TIR intracitoplasmático. TRIF recluta a TRAF3, una E3 ubiquitin ligasa, que junto con TRAF6 forman un andamio de poliubiquitina que servirá de anclaje a NEMO, TANK, IKKε y TBK1, favoreciendo la actividad quinasa de TBK1 que fosforilará a IRF3. Finalmente, IRF3 se transloca al núcleo y actuará como factor de transcripción de los genes de interferón tipo I.
- TLR-7 y TLR-9
El proceso es similar al del TLR4. El dominio TIR de MyD88 recluta al complejo IRAK1/IRAK4. IRAK1 puede asociarse fácilmente a IRF7, lo que hace que este quede fosforilado por la acción de IRAK1. Ahora IRAK1 actuará como factor de transcripción en el núcleo expresando los genes de interferón tipo I.
Son homodímeros. Son TLRs más hidrofílicos ya que reconocen ácidos nucleicos.
RIG-1 y MDA5
Cuando un virus intracelular penetra en la célula, este va a ser reconocido por una familia de proteínas denominadas receptores tipo RIG-I (RLR). Estos receptores se encuentran en el citoplasma de las células y no se encuentran anclados a la membrana. Su mecanismo de acción se basa en el reconocimiento de los virus RNA.
RIG-1 es capaz de discriminar entre el RNA viral y el propio, en función de las modificaciones postranscripcionales que presente dicho RNA.
Las tres posibles modificaciones post-transcripcionales son:
- RNA con un extremo trifosfato sin grupo 7 metil-guanosina en 5’
- Cap0: Este tipo de RNA va a tener un grupo 7 metil-guanosina en el extremo 5’
- Cap1: El RNA va a tener un grupo metilo en oxígeno 3’ del anillo de ribosa del primer nucleótido en 5’
- Cap2: El RNA va a tener un grupo metilo en el oxígeno 3’ del anillo de ribosa tanto en el primer como en el segundo nucleótido del extremo 5’.
Dado que la mayoría de los virus no se replican en el núcleo, qué es dónde ocurren las modificaciones que si afectan al RNA propio, RIG-1 podrá detectar los extremos 5’ trifosfato no metilados de los virus. En el caso de los picornavirus, que carecen de extremo 5’ trifosfato, RIG-1 no es capaz de reconocerlos.
Estructura de RIG-I
Presenta dos dominios CARD (caspase recruitment domain) que interaccionan con moléculas adaptadoras para la inducir la producción de interferon tipo I, así como dominios similares a helicasa mediante los cuales se une a las moléculas de RNA.

Mecanismo de acción
En un principio, RIG-I se va a encontrar autoinhibido por el plegamiento de su secuencia aminoacídica y estabilizado por interacciones entre los dominios CARD y helicasa.. El reconocimiento del RNA sin 7-metil-guanosina en 5’ o los RNA cap0 y cap1 gracias a la histidina 830 de su centro catalítico, va a provocar un cambio conformacional, alterando las interacciones que estabilizaban el plegamiento y haciendo qué RIG-1 se active y se despliegue.
Este proceso, va a dejar expuesto unos dominios CARD de RIG-I que les van a permitir interaccionar con los dominios CARD de unas proteínas mitocondriales llamadas MAVS. La interacción con MAVS va a mediar el reclutamiento de TRAFs que van a polimerizar una cadena de ubiquitina, que servirá como andamiaje a las IKKs, las cuales, una vez activadas, fosforilarán a:
- - IRF-3: que se dirigirá al núcleo y mediará la expresión de genes de interferón tipo
- - IkB: que será degradado vía proteasoma y permitirá que NFkB se transloque al núcleo y favorecerá la expresión de citoquinas proinflamatorias.
NOTA: Junto con RIG-I existen otras moléculas que realizan funciones similares como son:
- - MAD-5 (asociado a la diferenciación de melanoma tipo 5): también llamado helicard, es similar en estructura a RIG-I, pero detecta dsRNA (como el de los Picornavirus)
- - LGP2 (de la familia de RLR (RIG-like receptors)) contiene un dominio helicasa pero no CARD y se encarga de cooperar con RIG-I y MAD-5.
STING
STING (Stimulator of interferon genes) es una proteína anclada al retículo endoplasmático, orientada hacia el citosol. Esta estructura se va a encargar de reconocer moléculas procedentes tanto de virus como de bacterias. Su acción está mediada por la proteína cGAS, ya que reconoce dinucleótidos cíclicos (CDNs) como c-di-GMP o c-di-AMP que actúan como segundos mensajeros producidos por bacterias y virus:
- Virus: Los virus DNA secretan su material genético en la célula y este activa a la proteína cGAS (Cyclic GMP-AMP (cGAMP) synthase) (que se une a ellos a través de estructuras denominadas dedos de Zinc). cGAS activa se va a encargar de la formación de heterodinucleótidos de guanina y adenina que van a ser reconocidos por STING.
- Bacterias: Las bacterias van a secretar homodinucleótidos de guanina y adenina que van a ser también reconocidos por STING.
En ambos casos, el reconocimiento de los dinucleótidos por STING va a provocar la dimerización de la proteína y la activación de TBK1, que va a ser fosforilada. Trás esto, TBK1 fosforilará a IRF3 que se translocará al núcleo favoreciendo la expresión de genes de interferón tipo I.
NOD
Mientras que los TLRs son principalmente elementos sensores de productos microbianos extracelulares, los NOD (nucleotide-binding oligomerization domain) son receptores citoplasmáticos sensores de productos microbianos que se van a encontrar en el citoplasma.
Los NOD presentan un dominio de oligomerización de unión a nucleótidos, que va a ser el que favorezca la activación del mismo y la consecuente respuesta celular. Se han descubierto otras familias similares a NOD conocidas como NLR (NOD like receptors) que también intervienen en proceso de la respuesta innata.
Estructura de los NOD.
La estructura de los NOD va a estar constituida por dos dominios CARDs, el dominio NBD y un dominio LRR similar al de los TLRs. El dominio LRR es el que les va a permitir sensar esos productos microbianos creados por la bacteria.
A continuación recogemos una tabla resumen de los NOD más estudiados con sus ligandos:
Mecanismo de acción:
Una vez activado NOD por uno de sus ligandos antes mencionados en la tabla, se va a producir la dimerización de la proteína y el reclutamiento de RIP2 (Receptor-interacting serine/threonine-protein kinase 2) y XIAP (X-linked inhibitor of apoptosis protein) gracias a sus dominios CARD.
XIAP va a generar un andamio de poliubiquitina qué va a servir de estructura de anclaje para TAB1/2, TAK1 y NEMO junto con IKKα e IKKβ. TAK1 se activará por fosforilación, y tras esto fosforilará a IKKβ.
IKKβ favorecerá la degradación de IkB mediante su fosforilación, y permitirá la liberación de NFkB que se translocará al núcleo y favorecerá la expresión de citoquinas proinflamatorias.
Inflamasoma
El inflamasoma es un conjunto de moléculas que inducen la producción de citocinas proinflamatorias :IL-1β ,IL-18 e IL-33.
Constituidas por:
- - Una molécula sensora (NLR)
- - Otra adaptadora (ASC)
- - Otra efectora (caspasa 1).
La caspasa 1 requiere un procesamiento previo para actuar que se realiza en el inflamasoma. Su acción es escindir los propéptido de las citocinas proinflamatorias para generar las formas activas que se liberan al medio para ejercer su función. En este grupo se incluyen aquellas moléculas que presentan un dominio pirina, y se les conoce como la familia de los NLRP. A continuación veremos cuatro de las familias más estudiadas de los NLRP:
NLRP3
Reside de forma inactiva en el citoplasma, donde sus dominios LRR se unen a la chaperona HSP90 y a la co-chaperona SGT1. Existen unos determinados eventos que inducen la señalización por NLRP3:
- disminución de K+ intracelular → causa la disociación de HSP90 y SGT1.
- generación de ROS
- disrupción de lisosomas.
La disminución de K+ se da durante la infección debido a la formación de poros por toxinas de microorganismos patógenos como S.aureus. El modelo de señalización de NLRP3 mediado por ROS implica la oxidación de proteínas sensoras TRX (tiorredoxinas) que normalmente están unidas a TXNIP (thioredoxin-interacting protein) pero que al oxidarse se disocian, causando que la TXNIP libre desplace a la HSP90 y a SGT1, induciendo la activación. Por último, la fagocitosis de sustancias, como el adyuvante alum, una sal cristalina de aluminio, potasio y sulfatos, puede provocar la ruptura de lisosomas y la liberación de catepsina B activa, que estimula la vía NLRP3.
Mecanismo de acción:
NLRP3 induce la producción de citoquinas pro-inflamatorias y la muerte celular mediante un complejo multiproteico, el inflamasoma, que actúa en varias etapas.
El proceso se inicia con la agregación de dominios LRR de varias moléculas de NLRP3 en respuesta a un reconocimiento desencadenante. La agregación induce la interacción de dominios pirina con los dominios pirina de la proteína ASC (apoptosis-associated speck-like protein containing a carboxy-terminal CARD), una proteína adaptadora con un dominio pirina en el extremo aminoterminal y un dominio CARD en el extremo carboxiterminal.
La interacción causa la formación de un filamento ASC polimérico, que presenta el dominio pirina en el centro y los dominios CARD orientados hacia el exterior. Estos dominios CARD interaccionan con los dominios CARD de una proteasa inactiva, la pro-caspasa 1, iniciando un proceso de polimerización dependiente de CARD que da lugar a filamentos de caspasa 1
Está agregación desencadena el autoprocesamiento de la pro-caspasa 1, que libera el fragmento activo de caspasa 1, siendo ésta quien lleve a cabo el procesamiento proteolítico dependiente de ATP de citoquinas pro-inflamatorias, principalmente IL-1ꞵ y IL-18, en sus formas activas
La activación de caspasa 1 también induce una forma de muerte celular conocida como piroptosis, mediante un mecanismo desconocido aún.
NLRP1
Se da en monocitos y células dendríticas. Se activa por los productos de B. anthracis que va a secretar dos proteínas: factor letal (LF) y antígeno protectivo (PA). PA genera un poro en la membrana del hospedador que usa a LF para entrar en la célula.
- - Si hay altas dosis de LF se activará NLRP1 y se producirá la muerte por apoptosis.
- - Si las dosis son bajas, NLRP1 se activa y se genera IL-1β.
Existen dos teorías sobre la activación de NLRP1:
- - Activación directa: LF corta un fragmento aminoterminal y se activa.
- - Activación indirecta: Existe una proteína desconocida que inhibe NLRP1. Entonces LF actuaría escindiendo a la proteína desconocida y activando el dominio CARD.
NLRC4
NLRC4 se incluye en ocasiones dentro del grupo de molécula adaptadoras, porque actúa como adaptador con otras dos proteínas NLR: NAIP2 y NAIP5 (NLR Family Apoptosis Inhibitory Protein), qué detectan proteínas bacterianas que entran en la célula mediante sistemas de secreción usados por patógenos para acceder a nutrientes o transportar materiales desde la célula hospedadora.
La proteína PrgJ es componente del sistema de secreción tipo 3 (T3SS), un complejo macromolecular con forma de aguja.
Cuando se produce la infección por patógenos como Salmonella , PrgJ entra a la célula y es reconocida por NLRC4 y por NAIP2. La flagelina extracelular es reconocida por TLR5, pero dado que no entra con PrgJ por T3SS, es reconocida por NLRC4 en conjunción con NAIP5.
El flagelo y T3SS tienen una estructura similar: en la base la estructura es idéntica, y la zona de transición es similar, pero en el caso de T3SS el interior está formado por múltiples cadenas de proteína Rod, y acaba finalmente en la región aguja, qué es lo que realmente penetra en el citoplasma celular.
El sistema de inyección inyecta flagelina, proteína Rod y PrgJ, así como monómeros de la propia proteína de inyección, PrgI.
Estas proteínas serán las qué activen el sistema del inflamasoma.
En bacterias como Shigella, que carecen de flagelos, existe un plásmido que codifica para el sistema de inyección. En este caso, sólo se produce Rod y proteína aguja.
Al inyectar las proteínas, se adhieren cada una a su correspondiente proteína NAIP y reclutan a ASC mediante la formación de una estructura en forma de roseta, con dos anillos, uno interno y otro externo. Está estructura es similar a la de los TLR, con regiones ricas en leucina.
AIM
La familia AIM2 (absent in melanoma 2) es una familia incluida dentro de los inflamasomas que se activa por bacterias citosólicas y virus de DNA. Se caracteriza por contener el dominio HIN, que reconoce el DNA de cadena doble y desencadena la activación de la caspasa 1. AIM2 se localiza en el citoplasma. In vitro, se ha visto qué es importante en la respuesta frente a Vaccinia virus (VACV). In vivo, su deficiencia ha demostrado estar relacionada con una mayor susceptibilidad a infecciones por F. novicida. Tiene dos vías de activación:
- - Vía no canónica de activación: esta vía se da cuando las bacterias son fagocitadas, como en el caso de Francisella novicida, y escapan de las vacuolas, favoreciendo la producción y activación de cGAS, la activación de STING e IFI204 e inducen la producción de IFN tipo I. Este IFN tipo I, a través de unos receptores de INF tipo I, median la inducción de GBPs los cuales actúan sobre la vacuola y, después, sobre el propio contenido de la bacteria. Esto genera la liberación del DNA bacteriano y la activación de AIM2.
- - Vía canónica de activación: se da en el caso de virus (como MCMV) o de DNA transfectado. Este DNA es reconocido por AIM2 activándose.
AIM2 activo en ambos casos, recluta a ASC como molécula mediadora y activa a la caspasa 1 provocando o la piroptosis celular o bien la activación de la pro-IL-1β a IL-1β

Trabajo realizado por Iman L.B., Marcos M.B., Mª Marina M.V. & Alberto Manuel P.P.
